Usa R!
Una introducción a la programación
¿Qué es “programar”?
Programar es el proceso de crear un conjunto de instrucciones para decirle a la computadora cómo realizar una tarea.
¿Cómo programar?
La tarea de “programar” no se centra exclusivamente en escribir código, si no que conlleva una serie de pasos previos antes de “codificar” las instrucciones.
- Descomponer un problema complejo en partes más pequeñas.
- Reconocimiento de patrones repetitivos.
- Resolución de los problemas paso a paso.
- Escribir el código.
¿Qué es un “lenguaje de programación”?
Un lenguaje de programación es un lenguaje formal (es decir, un lenguaje con reglas gramaticales bien definidas) que proporciona al programador la capacidad y habilidad de escribir instrucciones o secuencias de órdenes para controlar el comportamiento de un sistema informático.
¿El entorno de R?
R es un conjunto integrado de software para la manipulación, cálculo y visualización de datos. Entre otras cosas tiene:
- Tratamiento y almacenamiento de datos eficaz.
- Conjunto de operadores para el calculo sobre matrices.
- Conjunto integrado de herramientas para el análisis y visualización de datos.
- Un simple y efectivo lenguaje de programación.
- Ampliamente utilizado en investigación cientifica.
¿Qué es un “Programa”?
Un programa informático es una secuencia de instrucciones basadas en un lenguaje de programación que el ordenador interpreta para resolver un problema.
¿Cuales son los elementos básicos de un programa?
- Instrucciones: conjunto de ordenes que indican al ordenador como realizar una tarea específica (asignar un valor a una variable, ejecutar un bucle que itera sobre una lista de datos).
- Funciones: conjunto de instrucciones que permiten realizar una tarea específica (ANOVA, plot de datos, etc..). Se pueden imaginar como una máquina que toma ciertos datos, realiza una operación y devuelve un resultado.
¿Cuales son los elementos básicos de un programa?
- Datos: pueden ser números, texto, imagénes, etc.. . En resumen cualquier tipo de información que el programa manipule. En general, son la razon de ser de muchas aplicaciones.
- Operadores: son las herramientas que permiten realizar operaciones sobre los datos (sumar, multiplicar, comparar, asignar, etc..).
- Variables: actuan como contenedores de información permitiendo que un programa conserve información y realice operaciones sobre esta información, en cierto modo se puede decir que son la “memoria” del programa.
“¡Hola Mundo!”
El programa “¡Hola Mundo!” suele ser el primer ejercicio típico en la introducción del estudio de un lenguaje de programación.
Usando funciones
- head()
Para ilustrar el uso de la función head() utilizaremos el dataset iris que viene con la instalación de R-Base.
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosaUsando funciones
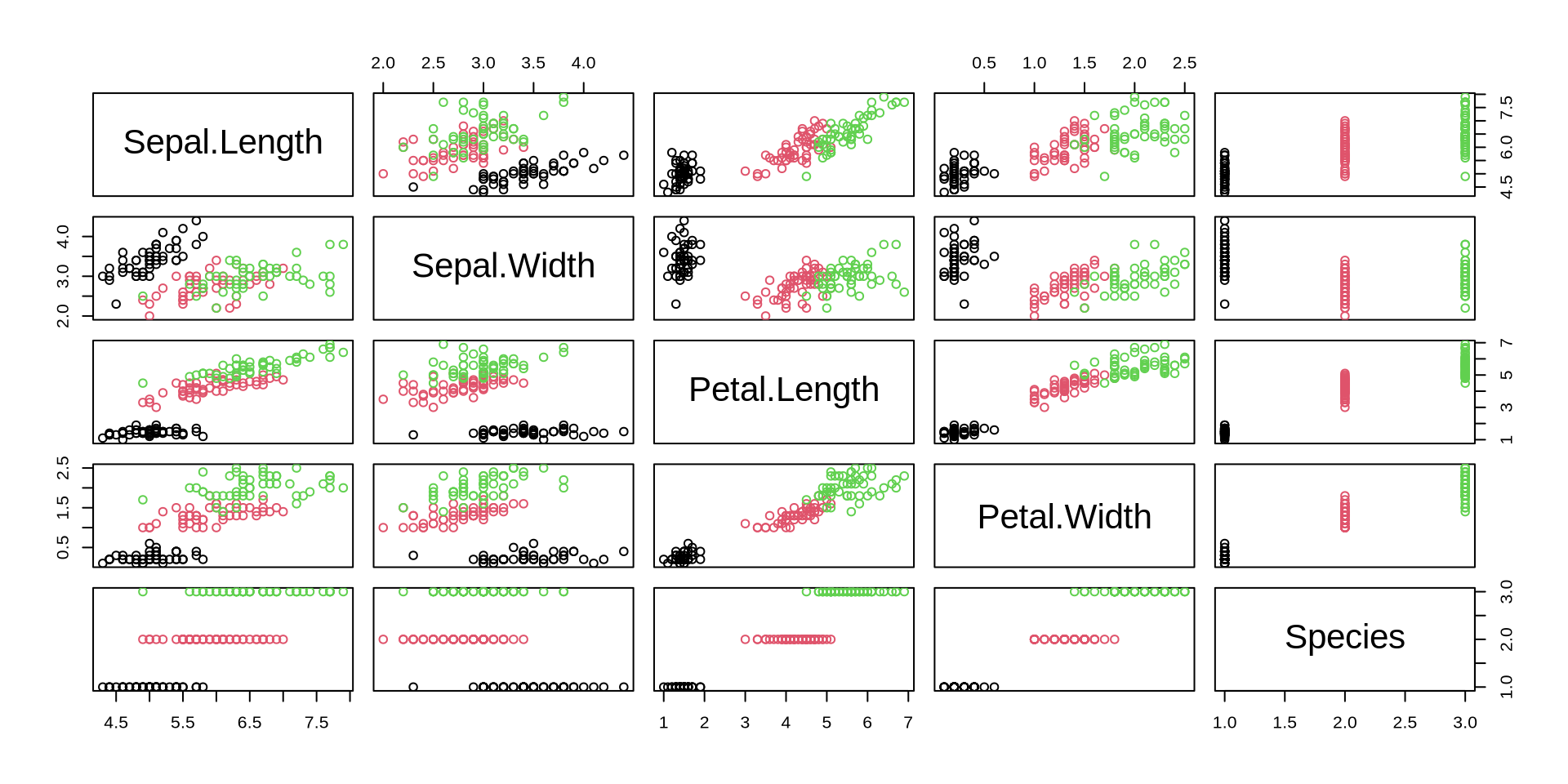
- pairs()
Para ilustrar el uso de la función pairs() utilizaremos el dataset iris que viene con la instalación de R-Base.
Estructuras de control de flujo
Se llaman estructuras de control de flujo a las instrucciones que permiten controlar las acciones de un algoritmo o programa. Estas son de gran utilidad para determinar la lógica y el orden en que ocurren las operaciones.
if, else: if(“si”) es usado cuando deseamos que una operación se ejecute únicamente cuando una condición se cumple. else(“de otro modo”) es usado para indicar que hacer en cado de que la condición de un if no se cumpla.
for: nos permite ejecutar un bucle, realizando una misma operación para cada elemento de un conjunto de datos.
if, else
[1] "la variable_1 NO es mayor que 10 "for
[1] "PRIMAVERA"
[1] "VERANO"
[1] "OTOÑO"
[1] "INVIERNO"Variables
Las variables actuan como contenedores de información (“memoria”) del programa. La estructura de esa información es muy heterogenea, podemos tener información simple(el número 5, el texto “¡Hola Mundo!”, etc..) o mucho más compleja (matriz de RNA-seq, con miles de filas y columnas).
En base al tipo de información que contienen las variables las podemos clasificar en simples o complejas.
Variables simples
| Tipo de dato | Descripción | Definición |
|---|---|---|
| Numeric | Números decimales | numero <- 1.0 |
| Integer | Números enteros | entero <- 1 |
| Character | Cadenas de texto | texto <- “un texto” |
| Complex | Números complejos | complejo <- 3 + 2i |
| Logical | TRUE o FALSE | 5 < 6; 5 == 5 |
| Factor | Es una variable de tipo categórica |
Variables complejas
La estructura de la información contenida en estas variables es más compleja. Además estas variables tienen métodos y atributos que facilitan acceder a la información que contienen.
Vectores: Los vectores almacena una secuencia de valores simples todos del mismo tipo.
Listas: A diferencia de los vectores las listas es una colección de elementos que pueden ser de diferente tipo.
Matrices:Una matriz es una estructura bidimensional que almacena números.
Data Frames: Un Data Frame es una estructura bidimensional que puede almacenar tipos de datos mixtos.
Vectores
La forma más habitual de crear un vector es usando la función c()
[1] 2 4 6 8 [1] 10 11 12 13 14 15 16 17 18 19 20 [1] 2 4 6 8 10 11 12 13 14 15 16 17 18 19 20Aritmética de Vectores
Las operaciones aritméticas con vectores se realizan posición a posición.
Aritmetica de Vectores
Dos vectores
Aritmetica de Vectores
Vectores texto y números
Elementos vector
Elementos vector
Máscara lógica
[1] 10 11 12 13 14 15 16 17 18 19 20 [1] TRUE TRUE TRUE TRUE TRUE FALSE FALSE FALSE FALSE FALSE FALSE[1] 10 11 12 13 14Conclusión
Para seleccionar subconjuntos de datos lo podemos realizar de dos formas:
- Vector de índices
[1] 890.54327 826.37310 251.42079 945.70646 598.31414 29.88844 310.83362
[8] 455.57611 602.91052 300.44098[1] 890.5433 598.3141 300.4410- Máscara lógica
[1] 251.42079 29.88844 310.83362 455.57611 300.44098Data Frame
Un data frame es una estructura de datos bidimensional pudiendo almacenar datos mixtos(texto, números, etc ..). Un data frame es una lista de vectores (columnas) de la misma longitd.
[1] 150 5 Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa Sepal.Length Sepal.Width Petal.Length Petal.Width
Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100
1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300
Median :5.800 Median :3.000 Median :4.350 Median :1.300
Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
Species
setosa :50
versicolor:50
virginica :50
Structure of an Arbitrary R Object
La función str() muestra la estructura interna de un Objeto(variable compleja) de R.
'data.frame': 150 obs. of 5 variables:
$ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
$ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
$ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
$ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...
$ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ... [1] 5.1 4.9 4.7 4.6 5.0 5.4 4.6 5.0 4.4 4.9 5.4 4.8 4.8 4.3 5.8 5.7 5.4 5.1
[19] 5.7 5.1 5.4 5.1 4.6 5.1 4.8 5.0 5.0 5.2 5.2 4.7 4.8 5.4 5.2 5.5 4.9 5.0
[37] 5.5 4.9 4.4 5.1 5.0 4.5 4.4 5.0 5.1 4.8 5.1 4.6 5.3 5.0 7.0 6.4 6.9 5.5
[55] 6.5 5.7 6.3 4.9 6.6 5.2 5.0 5.9 6.0 6.1 5.6 6.7 5.6 5.8 6.2 5.6 5.9 6.1
[73] 6.3 6.1 6.4 6.6 6.8 6.7 6.0 5.7 5.5 5.5 5.8 6.0 5.4 6.0 6.7 6.3 5.6 5.5
[91] 5.5 6.1 5.8 5.0 5.6 5.7 5.7 6.2 5.1 5.7 6.3 5.8 7.1 6.3 6.5 7.6 4.9 7.3
[109] 6.7 7.2 6.5 6.4 6.8 5.7 5.8 6.4 6.5 7.7 7.7 6.0 6.9 5.6 7.7 6.3 6.7 7.2
[127] 6.2 6.1 6.4 7.2 7.4 7.9 6.4 6.3 6.1 7.7 6.3 6.4 6.0 6.9 6.7 6.9 5.8 6.8
[145] 6.7 6.7 6.3 6.5 6.2 5.9Seleccionar una columna
[1] 5.1 4.9 4.7 4.6 5.0 5.4 4.6 5.0 4.4 4.9 5.4 4.8 4.8 4.3 5.8 5.7 5.4 5.1
[19] 5.7 5.1 5.4 5.1 4.6 5.1 4.8 5.0 5.0 5.2 5.2 4.7 4.8 5.4 5.2 5.5 4.9 5.0
[37] 5.5 4.9 4.4 5.1 5.0 4.5 4.4 5.0 5.1 4.8 5.1 4.6 5.3 5.0 7.0 6.4 6.9 5.5
[55] 6.5 5.7 6.3 4.9 6.6 5.2 5.0 5.9 6.0 6.1 5.6 6.7 5.6 5.8 6.2 5.6 5.9 6.1
[73] 6.3 6.1 6.4 6.6 6.8 6.7 6.0 5.7 5.5 5.5 5.8 6.0 5.4 6.0 6.7 6.3 5.6 5.5
[91] 5.5 6.1 5.8 5.0 5.6 5.7 5.7 6.2 5.1 5.7 6.3 5.8 7.1 6.3 6.5 7.6 4.9 7.3
[109] 6.7 7.2 6.5 6.4 6.8 5.7 5.8 6.4 6.5 7.7 7.7 6.0 6.9 5.6 7.7 6.3 6.7 7.2
[127] 6.2 6.1 6.4 7.2 7.4 7.9 6.4 6.3 6.1 7.7 6.3 6.4 6.0 6.9 6.7 6.9 5.8 6.8
[145] 6.7 6.7 6.3 6.5 6.2 5.9 [1] 5.1 4.9 4.7 4.6 5.0 5.4 4.6 5.0 4.4 4.9 5.4 4.8 4.8 4.3 5.8 5.7 5.4 5.1
[19] 5.7 5.1 5.4 5.1 4.6 5.1 4.8 5.0 5.0 5.2 5.2 4.7 4.8 5.4 5.2 5.5 4.9 5.0
[37] 5.5 4.9 4.4 5.1 5.0 4.5 4.4 5.0 5.1 4.8 5.1 4.6 5.3 5.0 7.0 6.4 6.9 5.5
[55] 6.5 5.7 6.3 4.9 6.6 5.2 5.0 5.9 6.0 6.1 5.6 6.7 5.6 5.8 6.2 5.6 5.9 6.1
[73] 6.3 6.1 6.4 6.6 6.8 6.7 6.0 5.7 5.5 5.5 5.8 6.0 5.4 6.0 6.7 6.3 5.6 5.5
[91] 5.5 6.1 5.8 5.0 5.6 5.7 5.7 6.2 5.1 5.7 6.3 5.8 7.1 6.3 6.5 7.6 4.9 7.3
[109] 6.7 7.2 6.5 6.4 6.8 5.7 5.8 6.4 6.5 7.7 7.7 6.0 6.9 5.6 7.7 6.3 6.7 7.2
[127] 6.2 6.1 6.4 7.2 7.4 7.9 6.4 6.3 6.1 7.7 6.3 6.4 6.0 6.9 6.7 6.9 5.8 6.8
[145] 6.7 6.7 6.3 6.5 6.2 5.9 [1] 5.1 4.9 4.7 4.6 5.0 5.4 4.6 5.0 4.4 4.9 5.4 4.8 4.8 4.3 5.8 5.7 5.4 5.1
[19] 5.7 5.1 5.4 5.1 4.6 5.1 4.8 5.0 5.0 5.2 5.2 4.7 4.8 5.4 5.2 5.5 4.9 5.0
[37] 5.5 4.9 4.4 5.1 5.0 4.5 4.4 5.0 5.1 4.8 5.1 4.6 5.3 5.0 7.0 6.4 6.9 5.5
[55] 6.5 5.7 6.3 4.9 6.6 5.2 5.0 5.9 6.0 6.1 5.6 6.7 5.6 5.8 6.2 5.6 5.9 6.1
[73] 6.3 6.1 6.4 6.6 6.8 6.7 6.0 5.7 5.5 5.5 5.8 6.0 5.4 6.0 6.7 6.3 5.6 5.5
[91] 5.5 6.1 5.8 5.0 5.6 5.7 5.7 6.2 5.1 5.7 6.3 5.8 7.1 6.3 6.5 7.6 4.9 7.3
[109] 6.7 7.2 6.5 6.4 6.8 5.7 5.8 6.4 6.5 7.7 7.7 6.0 6.9 5.6 7.7 6.3 6.7 7.2
[127] 6.2 6.1 6.4 7.2 7.4 7.9 6.4 6.3 6.1 7.7 6.3 6.4 6.0 6.9 6.7 6.9 5.8 6.8
[145] 6.7 6.7 6.3 6.5 6.2 5.9Seleccionar varias filas y columnas
Subset
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
51 7.0 3.2 4.7 1.4 versicolor
52 6.4 3.2 4.5 1.5 versicolor
53 6.9 3.1 4.9 1.5 versicolor
55 6.5 2.8 4.6 1.5 versicolor
57 6.3 3.3 4.7 1.6 versicolor
59 6.6 2.9 4.6 1.3 versicolor
64 6.1 2.9 4.7 1.4 versicolor
66 6.7 3.1 4.4 1.4 versicolor
69 6.2 2.2 4.5 1.5 versicolor
72 6.1 2.8 4.0 1.3 versicolor
73 6.3 2.5 4.9 1.5 versicolor
74 6.1 2.8 4.7 1.2 versicolor
75 6.4 2.9 4.3 1.3 versicolor
76 6.6 3.0 4.4 1.4 versicolor
77 6.8 2.8 4.8 1.4 versicolor
78 6.7 3.0 5.0 1.7 versicolor
87 6.7 3.1 4.7 1.5 versicolor
88 6.3 2.3 4.4 1.3 versicolor
92 6.1 3.0 4.6 1.4 versicolor
98 6.2 2.9 4.3 1.3 versicolorColumnas
Sepal.Length Sepal.Width Petal.Length Petal.Width Species Sepal.Ratio
1 5.1 3.5 1.4 0.2 setosa 1.457143
2 4.9 3.0 1.4 0.2 setosa 1.633333
3 4.7 3.2 1.3 0.2 setosa 1.468750
Petal.Ratio
1 7.0
2 7.0
3 6.5 Species Sepal.Ratio Petal.Ratio
1 setosa 1.457143 7.0
2 setosa 1.633333 7.0
3 setosa 1.468750 6.5Crear dataframe
Species Sepal.Ratio Petal.Ratio
1 setosa 1.457143 7.00
2 setosa 1.633333 7.00
3 setosa 1.468750 6.50
4 setosa 1.483871 7.50
5 setosa 1.388889 7.00
6 setosa 1.384615 4.25Función aggregate
La función aggregate() es muy util para calcular estadisticos por subconjuntos del data frame.
Species Sepal.Length Sepal.Width Petal.Length Petal.Width
1 setosa 5.006 3.428 1.462 0.246
2 versicolor 5.936 2.770 4.260 1.326
3 virginica 6.588 2.974 5.552 2.026Drosophila melanogaster
Para el análisis de expresión diferencial vamos a utilizar el paquete DESeq2.
Debemos familiarizarnos con la función a usar:
La función DESeq() recibe como paramatro un objeto de la clase DESeqDataSet. Este tipo de variable almacena los valores de entrada, calculos intermedios y resultados del análisis.
- countData: Es una matriz númerica (muestras en columnas).
- colData: DataFrame con al menos una columna, las filas corresponden a las columnas del countData.
- design: Expresa como las cuentas para cada gen dependen de las variables en colData.
Drosophila Melanogaster
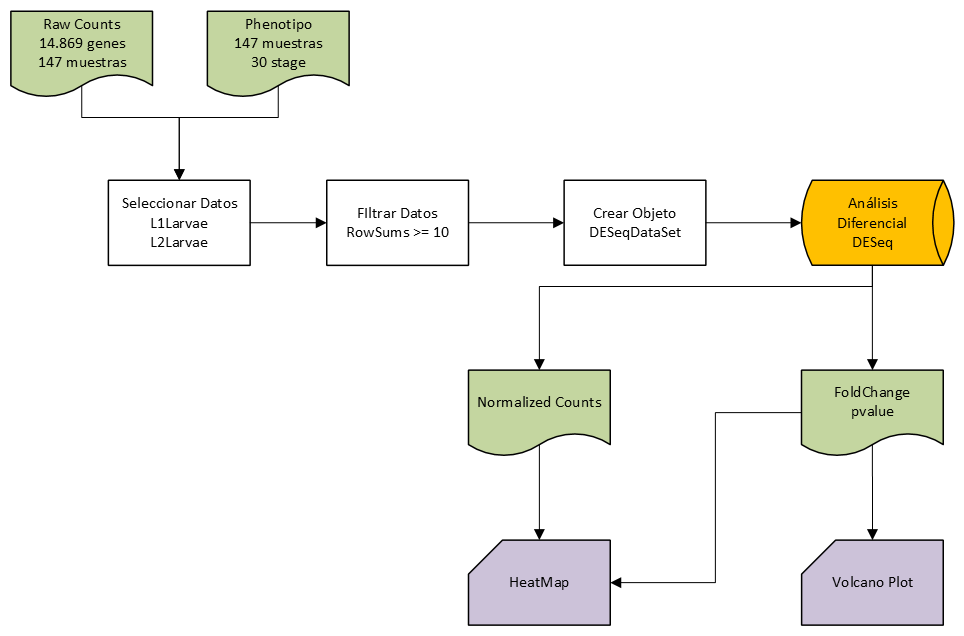
Raw counts
[1] 14869 148 gene SRX007811 SRX008180 SRX008227 SRX008238 SRX008258 SRX008015
1 FBgn0000003 0 0 0 0 0 0
2 FBgn0000008 3903 1601 681 648 718 921
3 FBgn0000014 75 29 10 9 12 563
4 FBgn0000015 33 11 8 4 8 235
5 FBgn0000017 9763 4107 1814 1652 1788 6358
6 FBgn0000018 832 368 146 126 170 97
SRX008179 SRX008190 SRX008193 SRX008271 SRX008027 SRX008181 SRX008217
1 0 0 0 0 0 0 0
2 295 364 268 267 1764 411 284
3 186 239 152 191 4890 957 806
4 95 101 58 101 3000 590 492
5 2214 2763 1906 2053 6006 1252 904
6 33 55 25 22 339 57 60
SRX008250 SRX008265 SRX008025 SRX008175 SRX008210 SRX008212 SRX008257
1 0 0 0 0 0 0 0
2 396 706 1074 563 156 160 440
3 1036 1956 6147 3166 956 1053 2876
4 715 1185 2492 1228 340 458 1174
5 1244 2456 5030 2584 746 847 2105
6 71 116 178 95 34 35 75
SRX008010 SRX008249 SRX008252 SRX008273 SRX008274 SRX008005 SRX008198
1 0 0 0 0 0 0 0
2 1557 874 904 632 234 4488 1222
3 5160 3270 2836 2048 815 4792 1390
4 1986 1454 1138 788 324 1709 541
5 5350 3101 3046 2343 969 8695 2448
6 130 99 75 56 25 141 43
SRX008208 SRX008243 SRX008247 SRX008018 SRX008177 SRX008225 SRX008235
1 0 0 0 0 0 0 0
2 825 3030 1224 8755 2431 1718 5478
3 883 3102 1192 9943 2536 1890 5782
4 370 1076 431 3421 886 744 1942
5 1518 5540 2345 12295 3296 2440 7652
6 25 97 34 229 59 50 113
SRX008277 SRX008007 SRX008196 SRX008233 SRX008237 SRX008262 SRX008006
1 0 0 0 0 0 0 0
2 2212 5617 1039 2769 1716 1664 1749
3 2559 5134 935 2339 1404 1426 2908
4 985 1313 240 645 392 419 1024
5 3125 9374 1816 4564 2640 2853 4548
6 48 122 19 67 27 44 93
SRX008178 SRX008205 SRX008242 SRX008278 SRX008020 SRX008213 SRX008215
1 0 0 0 0 0 0 0
2 1532 549 715 390 1685 384 1397
3 2602 965 1116 638 2333 543 1832
4 845 321 398 216 914 199 667
5 4156 1537 1816 1085 2577 572 2010
6 57 20 23 10 42 18 31
SRX008222 SRX008259 SRX008011 SRX008214 SRX008221 SRX008241 SRX008256
1 0 0 0 0 0 0 0
2 407 2141 1036 417 353 560 394
3 512 3101 1068 448 338 589 475
4 217 1153 419 165 139 219 179
5 526 3328 3327 1425 1046 1719 1342
6 12 72 35 16 11 15 15
SRX008019 SRX008167 SRX008234 SRX008251 SRX008266 SRX008026 SRX008174
1 0 0 0 0 0 0 0
2 2548 730 687 786 587 928 392
3 3090 979 819 1008 810 1845 796
4 1306 404 334 450 310 635 295
5 6979 1985 1843 2297 1539 1440 670
6 113 33 26 39 23 150 64
SRX008201 SRX008239 SRX008008 SRX008168 SRX008211 SRX008255 SRX008261
1 0 0 0 0 0 0 0
2 575 851 1696 353 284 733 258
3 1203 1740 2030 422 375 914 296
4 485 617 695 155 130 350 95
5 1021 1330 2360 481 434 1054 381
6 102 139 371 94 66 162 49
SRX008009 SRX008194 SRX008207 SRX008224 SRX008244 SRX008029 SRX008184
1 0 0 0 0 0 0 0
2 311 94 108 110 148 228 74
3 536 182 211 196 296 535 181
4 190 71 70 81 111 127 56
5 481 154 197 197 292 667 226
6 83 29 27 35 48 131 63
SRX008206 SRX008220 SRX008267 SRX008014 SRX008182 SRX008187 SRX008189
1 0 0 0 0 0 0 0
2 115 77 74 393 541 168 161
3 278 212 173 822 1342 360 434
4 62 56 44 166 324 82 95
5 357 242 216 1036 1416 490 452
6 82 48 57 107 167 44 57
SRX008200 SRX008156 SRX008199 SRX008245 SRX008253 SRX008023 SRX008218
1 0 0 0 0 0 0 0
2 163 1205 2939 945 1022 3602 926
3 398 1598 3795 1136 1349 3227 836
4 103 389 1029 322 338 470 134
5 423 1791 4400 1395 1484 5073 1312
6 39 154 334 94 124 307 78
SRX008246 SRX008248 SRX008275 SRX008016 SRX008192 SRX008219 SRX008236
1 0 0 0 0 0 0 0
2 2209 1494 1076 4011 1233 1758 2123
3 1903 1231 878 2698 885 1115 1577
4 287 201 130 529 192 229 325
5 3019 1972 1521 5793 1860 2517 3176
6 178 105 80 274 95 128 156
SRX008264 SRX008013 SRX008188 SRX008197 SRX008228 SRX008022 SRX008183
1 0 0 0 0 0 0 0
2 2023 1117 1004 1447 1452 665 396
3 1488 282 286 355 379 61 44
4 316 99 87 153 101 17 19
5 2960 2356 2159 3069 3182 2170 1317
6 148 264 247 320 333 218 149
SRX008185 SRX008216 SRX012269 SRX008024 SRX008173 SRX008195 SRX008202
1 0 0 0 0 0 0 0
2 686 409 1011 603 336 380 431
3 64 36 72 39 35 40 36
4 17 15 20 15 17 20 8
5 2252 1426 3357 1906 1123 1239 1551
6 220 137 343 194 138 164 143
SRX008254 SRX012270 SRX008012 SRX008170 SRX008263 SRX008268 SRX016332
1 0 0 0 0 0 0 0
2 437 738 717 630 389 303 373
3 29 54 340 307 195 148 222
4 10 12 256 270 165 116 142
5 1410 2504 1388 1279 790 579 849
6 136 224 170 186 107 76 108
SRX008028 SRX008191 SRX008223 SRX008260 SRX008021 SRX008203 SRX008226
1 0 0 0 0 0 0 0
2 1234 1112 989 1026 430 200 158
3 309 298 243 281 137 75 47
4 347 292 305 328 167 118 70
5 2007 1837 1596 1644 898 461 341
6 319 280 251 308 154 98 54
SRX008229 SRX008232 SRX012271 SRX008171 SRX008186 SRX008240 SRX010758
1 0 0 0 0 0 0 0
2 430 250 1074 3386 6313 3210 4811
3 132 74 339 647 1310 632 939
4 177 98 322 202 375 203 259
5 777 504 2100 2759 5211 2524 3931
6 137 71 332 128 263 96 165
SRX016331 SRX008155 SRX008169 SRX008172 SRX008231 SRX008542 SRX008017
1 0 0 0 0 0 0 0
2 3803 2393 1834 1890 744 952 971
3 751 572 404 440 156 212 307
4 219 252 183 210 76 81 146
5 3212 2584 2013 2156 809 963 1168
6 117 216 142 143 47 65 134
SRX008209 SRX008230 SRX008270 SRX008157 SRX008204 SRX008269 SRX008272
1 0 0 0 0 0 0 0
2 844 1204 620 1519 1267 1448 1185
3 253 371 159 1841 1650 1875 1370
4 146 168 93 340 332 368 261
5 1085 1513 782 1973 1592 1833 1465
6 124 146 82 101 112 116 102
SRX008276
1 0
2 3225
3 4048
4 711
5 4066
6 294Metadatos
[1] 147 3 sample.id num.tech.reps stage
1 SRX007811 5 Embryos0002
2 SRX008180 2 Embryos0002
3 SRX008227 1 Embryos0002
4 SRX008238 1 Embryos0002
5 SRX008258 1 Embryos0002
6 SRX008015 2 Embryos0204
7 SRX008179 1 Embryos0204
8 SRX008190 1 Embryos0204
9 SRX008193 1 Embryos0204
10 SRX008271 1 Embryos0204
11 SRX008027 4 Embryos0406
12 SRX008181 1 Embryos0406
13 SRX008217 1 Embryos0406
14 SRX008250 1 Embryos0406
15 SRX008265 2 Embryos0406
16 SRX008025 5 Embryos0608
17 SRX008175 2 Embryos0608
18 SRX008210 1 Embryos0608
19 SRX008212 1 Embryos0608
20 SRX008257 2 Embryos0608Fenotipos seleccionados
Queremos seleccionar las muestras que corresponden con el stage ‘L1Larvae’ y ‘L2Larvae’.
sample.id stage
61 SRX008026 L1Larvae
62 SRX008174 L1Larvae
63 SRX008201 L1Larvae
64 SRX008239 L1Larvae
65 SRX008008 L2Larvae
66 SRX008168 L2Larvae
67 SRX008211 L2Larvae
68 SRX008255 L2Larvae
69 SRX008261 L2LarvaeFiltrar el dataset por las muestras seleccionadas
[1] "gene" "SRX007811" "SRX008180" "SRX008227" "SRX008238" "SRX008258"
[7] "SRX008015" "SRX008179" "SRX008190" "SRX008193" "SRX008271" "SRX008027"
[13] "SRX008181" "SRX008217" "SRX008250" SRX008026 SRX008174 SRX008201 SRX008239 SRX008008 SRX008168 SRX008211
1 0 0 0 0 0 0 0
2 928 392 575 851 1696 353 284
3 1845 796 1203 1740 2030 422 375
4 635 295 485 617 695 155 130
5 1440 670 1021 1330 2360 481 434
6 150 64 102 139 371 94 66
SRX008255 SRX008261
1 0 0
2 733 258
3 914 296
4 350 95
5 1054 381
6 162 49Añadir genes
SRX008026 SRX008174 SRX008201 SRX008239 SRX008008 SRX008168
FBgn0000003 0 0 0 0 0 0
FBgn0000008 928 392 575 851 1696 353
FBgn0000014 1845 796 1203 1740 2030 422
FBgn0000015 635 295 485 617 695 155
FBgn0000017 1440 670 1021 1330 2360 481
FBgn0000018 150 64 102 139 371 94
SRX008211 SRX008255 SRX008261
FBgn0000003 0 0 0
FBgn0000008 284 733 258
FBgn0000014 375 914 296
FBgn0000015 130 350 95
FBgn0000017 434 1054 381
FBgn0000018 66 162 49TIP
Eliminar genes con pocas counts
FBgn0000003 FBgn0000008 FBgn0000014 FBgn0000015 FBgn0000017 FBgn0000018
0 6070 9621 3457 9171 1197 FBgn0000003 FBgn0000008 FBgn0000014 FBgn0000015 FBgn0000017 FBgn0000018
FALSE TRUE TRUE TRUE TRUE TRUE SRX008026 SRX008174 SRX008201 SRX008239 SRX008008 SRX008168
FBgn0000008 928 392 575 851 1696 353
FBgn0000014 1845 796 1203 1740 2030 422
FBgn0000015 635 295 485 617 695 155
FBgn0000017 1440 670 1021 1330 2360 481
FBgn0000018 150 64 102 139 371 94
FBgn0000024 1797 707 1126 1498 1701 341
SRX008211 SRX008255 SRX008261
FBgn0000008 284 733 258
FBgn0000014 375 914 296
FBgn0000015 130 350 95
FBgn0000017 434 1054 381
FBgn0000018 66 162 49
FBgn0000024 334 791 235[1] 11342 9DESeq2
Para el análisis de expresión diferencial vamos a utilizar el paquete DESeq2.
Vignettes: Analyzing RNA-seq data with DESeq2
DESeq2
stage
SRX008026 L1Larvae
SRX008174 L1Larvae
SRX008201 L1Larvae
SRX008239 L1Larvae
SRX008008 L2Larvae
SRX008168 L2Larvaeclass: DESeqDataSet
dim: 11342 9
metadata(1): version
assays(1): counts
rownames(11342): FBgn0000008 FBgn0000014 ... FBgn0261574 FBgn0261575
rowData names(0):
colnames(9): SRX008026 SRX008174 ... SRX008255 SRX008261
colData names(1): stageDESeq2
class: DESeqDataSet
dim: 11342 9
metadata(1): version
assays(4): counts mu H cooks
rownames(11342): FBgn0000008 FBgn0000014 ... FBgn0261574 FBgn0261575
rowData names(22): baseMean baseVar ... deviance maxCooks
colnames(9): SRX008026 SRX008174 ... SRX008255 SRX008261
colData names(2): stage sizeFactorlog2 fold change (MLE): stage L1Larvae vs L2Larvae
Wald test p-value: stage L1Larvae vs L2Larvae
DataFrame with 11342 rows and 6 columns
baseMean log2FoldChange lfcSE stat pvalue
<numeric> <numeric> <numeric> <numeric> <numeric>
FBgn0000008 562.467 0.0567848 0.0548497 1.03528 3.00538e-01
FBgn0000014 894.891 0.7973618 0.0453627 17.57748 3.66521e-69
FBgn0000015 323.316 0.8453126 0.0737835 11.45666 2.17741e-30
FBgn0000017 862.122 0.2538049 0.0478014 5.30957 1.09886e-07
FBgn0000018 111.781 -0.3815511 0.1121942 -3.40081 6.71867e-04
... ... ... ... ... ...
FBgn0261570 1415.4046 0.215624 0.0667683 3.22944 1.24034e-03
FBgn0261572 71.9698 2.201588 0.1526436 14.42307 3.70504e-47
FBgn0261573 311.1002 0.311384 0.0678953 4.58624 4.51307e-06
FBgn0261574 1477.7729 0.557713 0.0445722 12.51255 6.37406e-36
FBgn0261575 1214.1409 -0.346767 0.0481108 -7.20769 5.69101e-13
padj
<numeric>
FBgn0000008 3.49975e-01
FBgn0000014 2.56904e-68
FBgn0000015 8.69814e-30
FBgn0000017 2.25929e-07
FBgn0000018 1.08511e-03
... ...
FBgn0261570 1.95915e-03
FBgn0261572 1.96258e-46
FBgn0261573 8.46415e-06
FBgn0261574 2.79645e-35
FBgn0261575 1.45364e-12Guardar resultados
Guardamos los resultados del análisis diferencial y los datos normalizados